



suivant: Version 1D du modèle
monter: main
précédent: Entrées/Sorties
Table des matières
Sous-sections
Attention manuel martien. Se reporter a la page WEB de LMDZ4
Ce chapitre est destiné aux personnes n'ayant jamais utilisé le
modèle du LMD. Le meilleur moyen de prendre contact étant sans
doute de réaliser une simulation, voici la marche à suivre. A
partir de cet exemple, vous pourrez ensuite modifier à votre gré aussi
bien les paramètres de contrôle du run que les conditions initiales.
Vous trouverez dans le répertoire tous les fichiers
nécessaires à l'installation, la compilation et l'execution du
modèle .
- - Recopiez chez vous le répertoire de base du modèle
stocké dans (Son contenu est décrit section 5).
- - Affectez les variables d'environnement pour le modèle:
- LMDGCM Path du répertoire où vous avez installé
le modèle (Path complet de )
setenv LMDGCM $PATH1/LMDZ.MARS
- LIBOGCM Path du répertoire (ce répertoire est à créer:
libo par exemple) dans lequel seront
stockées les librairies objets créées lors de la compilation du
modèle.
setenv LIBOGCM $PATH2/libo
- - Installez NetCDF
et affectez les variables
d'environnement NCDFINC et NCDFLIB:
- La dernière version du package NetCDF est disponible sur le web à
l'adresse suivante:.
- Elle contient à la fois de quoi créer la librairie objet libnetcdf.a, les fichiers includes nécessaires, et de quoi compiler les logiciels NetCDF de base, ncdump et ncgen.
- Afin que le modèle, lors de la compilation, trouve les librairies et
includes correspondant au type de machine utilisée, il convient de déclarer
les variables d'environnement NCDFINC et NCDFLIB.
- NCDFLIB Nom du répertoire contenant la librairie objet (libnetcdf.a)
et NCDFINC nom du répertoire contenant les fichiers include (netcdf.inc) NetCDF pour les différentes plateformes.
setenv NCDFINC $PATH3/include
setenv NCDFLIB $PATH3/lib
- - Installez GrAdS
- Pour les gens travaillant au LMD, il suffit, grâce au brillantissime
Laurent Fairhead, pour avoir accès à GrAdS et à son environnement,
d'ajouter dans son .cshrc (c'est à priori fait dans .env_soft):
source /distrib/local/grads/grads.env
- - Recopiez makegcm le script pour la compilation (dans
$HOME/bin par exemple). Ce script est disponible dans
(Ou demander au LMD).
- - Enfin, assurez vous que vous avez bien accès à
tous les executables nécessaires pour l'utilisation du modèle.
Pensez a indiquer le chemin correspondant dans le PATH.
- - fonction UNIX make
- - le compilateur Fortran f90
- - ncdump
- - grads
Compiler le modèle
- Pour compiler par exemple le modèle martien en résolution
64x48x25, tapez
(conformément au manuel de la fonction makegcm décrit section 5.4)
makegcm -d 64x48x25 -p mars gcm
On récupère l'executable gcm.e (le modèle compilé) dans le
répertoire où on a lancé la commande makegcm.
- Autre exemple: Pour compiler le modèle martien avec 2 traceurs :
makegcm -d 64x48x25 -t 2 -p mars gcm
- Autre exemple:
Pour compiler le modèle martien pour vérifier les dépassement de
tableaux (debuggage : attention, le modèle est alors très lent !) :
makegcm -d 64x48x25 -p mars -O "-C" gcm
- Récuperez dans le répertoire $PATH1/LMDZ.MARS/deftank
le fichier
de paramètres run.def (décrits section
6.2) nécessaire au modèle. Récupérez en plus les
fichiers callphys.def et esasig.def OU z2sig.def
pour le modèle martien.
Les paramètres sont affectés par des valeurs
usuelles, et il est conseillé de réaliser une première simulation
sans les modifier.
- Recopiez les fichiers start et startfi (décrits section
6.2)
stockés au LMD (demander) ou créer ces fichiers à partir d'une ``banque
d'états initiaux start_archive (cf. section 7.8).
Ils contiennent des
conditions initiales de scénario standard et permettront de valider
l'installation du modèle.
Placez dans un même répertoire le programme gcm.e et les
fichiers d'entrée, puis tapez:
gcm.e
Si vous n'avez jamais utilisé le logiciel graphique GrAds, il
est vivement conseillé de consacrer une petite demi-heure à se
familiariser avec lui en effectuant la demonstration prévue à cet
effet. Facile, rapide, elle permet d'utiliser les commandes de base.
Pour cela, allez lire le fichier
/distrib/local/grads/sample
Prenons l'exemple de la visualisation des fichiers histmoy, histphy.
Les fichiers NetCDF histmoy.nc, histphy.nc... sont directement exploitables
sous GrAdS grâce à l'utilitaire gradsnc, dont l'appel est invisible à
l'utilisateur.
Pour visualiser par exemple la carte de la température dans la 5ème couche
à partir du fichier histmoy:
- Visualisation sous GrAdS:
grads return
- return (pour ouvrir une fenêtre landscape)
ga-> sdfopen histmoy.nc
ga-> query file (display des infos sur le fichier ouvert,
dont le nom des variables stockées. Racourcis: q file)
ga-> set z 5 (fixe l'altitude à la 5ème couche)
ga-> set t 1 (fixe le temps à la première valeur
stockée)
ga-> query dims (Indique les valeurs fixées pour les 4
dimensions. Racourcis: q dims)
ga-> display temp (display la carte de température pour la 5ème
couche et pour la première valeur de temps stockée. Raccourcis: d
T)
ga-> clear (pour effacer l'écran. Raccourcis: c)
ga-> set gxout shaded (mode d'affichage plein)
ga-> display temp
ga-> set gxout contour (retour au mode contour pour
afficher les niveaux)
ga-> display temp (superpose les contours si on ne repasse pas
par la commande clear)
A la fin d'une simulation, le modèle génère des fichiers
restart qui contiennent l'état final du modèle. Ces fichiers
( au même format que les fichiers start) peuvent donc servir de
conditions initiales à une nouvelle simulation.
Il suffit de les renommer:
mv restart.nc start.nc
mv restartfi.nc startfi.nc
et de relancer la simulation.
Dans la pratique, il est recommandé d'enchainer une série de simulations
de quelques jours ou quelques dizaines de jours (ou centaines à basse
résolution).
Pour cela, un script nommé run0 est disponible dans
$PATH1/LMDZ.MARS/deftank
Utilisation :
- Régler la durée de chaque simulation dans run.def
- Régler le nombre max de simulation dans l'entête de run0
- Recopier les fichiers de démarrage start.nc startfi.nc
sous le nom start0.nc startfi0.nc.
- Taper : run0
run0 lancera ainsi une série de simulations qui génèreront des
fichiers de sortie indéxés (e.g. start1, startfi1, diagfi1, etc..)
dont les fichiers lrun1, lrun2, etc. contenant la redirection de
l'écran avec les informations sur le déroulement du run.
NOTA: pour redémarrer une série de simulations après une première
série (par exemple pour partir de start5 et startfi5), il faut
simplement inscrire l'indice des fichiers initiaux (e.g. 5) dans le
fichier nommé num_run. Si num_run existe, le modèle démarrera
de l'indice contenu dans num_run. Sinon, il demarre de start0
et startfi0.
NOTA: Un script est disponible pour effectuer
des runs annuels avec 12 saisons de 30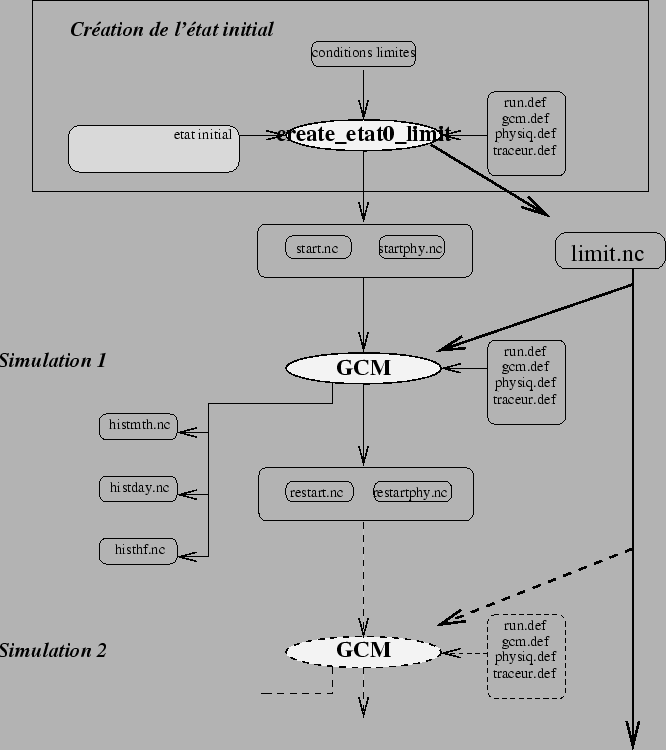 en longitude
solaire comme pour la base de données (script run_mcd dans le répertoire deftank également).
Ce script fonctionne avec le script run0. Il suffit de régler le nombre de simulation à 1 dans run0. Il faut recopier run.def en run.def.ref et mettre nday à 9999 dans ce dernier fichier. Il faut commenter les N mois dejà effectués et écrire N dans num_run si l'on veut rédémarrer à partir de startN.c
On lance ensuite run_mcd.
en longitude
solaire comme pour la base de données (script run_mcd dans le répertoire deftank également).
Ce script fonctionne avec le script run0. Il suffit de régler le nombre de simulation à 1 dans run0. Il faut recopier run.def en run.def.ref et mettre nday à 9999 dans ce dernier fichier. Il faut commenter les N mois dejà effectués et écrire N dans num_run si l'on veut rédémarrer à partir de startN.c
On lance ensuite run_mcd.
De nombreux paramètres du modèles (par exemple, la profondeur optique de
la poussière) sont stockés dans les états initiaux (fichiers
NetCDF start et startfi ).
Pour changer ces paramètres, ou plus généralement changer la résolution
du modèle, il faut utiliser le programme newstart.
Ce programme est aussi utilisé pour créer un état initial. En pratique,
dans la plupart des cas, on reprend un ancien état initial que l'on
modifie avec newstart .
le programme newstart se compile comme le GCM à la résolution
voulue. Par exemple :
makegcm -d 64x48x25 -p mars newstart
Il suffit ensuite de lancer
newstart.e
Le programme donne alors deux options :
A partir de quoi souhaitez vous creer vos etats initiaux ?
0 - d un fichier start_archive
1 - d un fichier start
- - L'option ``1'' permet de modifier des fichiers
start.nc, startfi.nc
- - L'option ``0'' permet de de lire les informations necessaires
à la création d'un nouvel état initial à partir du fichier
start_archive.nc .
Il suffit ensuite de répondre aux questions du menu déroulant.
Le programme newstart.e crée les fichiers
newstart.nc , newstartfi.nc
, qu'il faut généralement renommer.
Le fichier archive
start_archive.nc est crée à partir des fichiers
start.nc, startfi.nc par le programme start2archive.
Le programme start2archive se compile comme le GCM à la résolution
voulue. Par exemple :
makegcm -d 64x48x25 -p mars start2archive
Il suffit ensuite de lancer start2archive.e
Exemple, pour créer des états initiaux à la résolution
32 24
24 25 à partir de fichiers
NetCDF
25 à partir de fichiers
NetCDF start et startfi à la résolution
64 48
48 32 :
32 :
Un processus équivalent permet de changer le nombre de traceurs, de
créer un état initial pour un zoom, etc...




suivant: Version 1D du modèle
monter: main
précédent: Entrées/Sorties
Table des matières
Compte generique LMDZ
2004-04-05
 en longitude
solaire comme pour la base de données (script run_mcd dans le répertoire deftank également).
Ce script fonctionne avec le script run0. Il suffit de régler le nombre de simulation à 1 dans run0. Il faut recopier run.def en run.def.ref et mettre nday à 9999 dans ce dernier fichier. Il faut commenter les N mois dejà effectués et écrire N dans num_run si l'on veut rédémarrer à partir de startN.c
On lance ensuite run_mcd.
en longitude
solaire comme pour la base de données (script run_mcd dans le répertoire deftank également).
Ce script fonctionne avec le script run0. Il suffit de régler le nombre de simulation à 1 dans run0. Il faut recopier run.def en run.def.ref et mettre nday à 9999 dans ce dernier fichier. Il faut commenter les N mois dejà effectués et écrire N dans num_run si l'on veut rédémarrer à partir de startN.c
On lance ensuite run_mcd.